
Bioquímica General y Bucal - FOUBA
1
NUCLEÓTIDOS
Y
ÁCIDOS
NUCLEICOS
BIOSÍNTESIS
DE
ÁCIDOS
NUCLEICOS
Y
DE
PROTEÍNAS
INTRODUCCIÓN:
De entre las muchas moléculas biológicas que encierra la célula, en este capítulo serán de interés el
ADN (ácido desoxirribonucleico), el ARN (ácido ribonucleico) y las proteínas. Todas son
macromoléculas, es decir, moléculas de gran tamaño que forman polímeros lineales construidos a partir
de unidades simples o monómeros – para ADN y ARN son
NUCLEOTIDOS y para proteínas son
AMINOACIDOS. Los procesos que permiten mantener y perpetuar la vida derivan de la información
genética que 1) se transmite de una generación a la siguiente a través de la duplicación del ADN, 2) se
expresa a través de la transcripción o síntesis de ARN y la traducción o síntesis de proteínas y 3) a
veces mejora mediante la reparación del ADN y la recombinación génica. En todos estos procesos la
información de una secuencia lineal se utiliza para especificar otra cadena lineal.
La información genética total de un organismo se denomina genoma. La doble hélice se asocia a
proteínas ubicadas en el núcleo celular, llamadas histonas para formar una unidad proteína-ADN
llamada nucleosoma. Al conjunto de nucleosomas, que se asemeja a una cadena de cuentas de collar,
se lo denomina cromatina. Cuando la célula eucariota procede a la duplicación, todo el material
genético contenido en la doble hélice de ADN, organizado en cromatina, comienza a condensarse para
formar un empaquetamiento conocido como cromosomas (Fig. 1).
Fig. 1: Empaquetamiento del ADN con proteínas para formar la cromatina (Interfase). Nucleosomas compactados
para formar un cromosoma metafásico (Mitosis o Meiosis).

Bioquímica General y Bucal - FOUBA
2
El genoma humano contiene, aproximadamente, 6x10
9
pares de nucleótidos distribuidos en 22 pares
de cromosomas autosómicos y 2 cromosomas sexuales. Por lo tanto el conjunto de los 46 cromosomas
de una célula humana típica vistos al microscopio se denomina cariotipo humano. Este resulta de
utilidad en pruebas de filiación y en detecciones precoces de enfermedades provocadas por mala
segregación durante la formación de las gametas (meiosis).
ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS:
La unidad fundamental que los constituye se denomina nucleótido.
En general todo nucleótido está formado por tres componentes:
1.
una base nitrogenada heterocíclica
2.
una pentosa
3.
uno, dos o tres grupo/s fosfato/s
1) Bases nitrogenadas:
Son compuestos aromáticos heterocíclicos que derivan de la pirimidina (que contiene en un heterociclo
dos átomos de nitrógeno) o de la purina (dos heterociclos condensados con cuatro átomos de
nitrógeno) (Fig.2). Reciben el nombre de bases pirimidínicas la
citosina, timina (exclusiva del ADN) y
uracilo (exclusiva del ARN) y de bases púricas la adenina y guanina (Fig. 2).
Fig.2: Estructura de la pirimidina y de la purina. Bases pirimidínicas y bases púricas
2) Pentosas:
La ribosa está presente en los ribonucleótidos, en cambio la 2-desoxirribosa en los
desoxirribonucleótidos (Fig. 3). Los átomos de las pentosas se numeran como 1’,2’,3’, etc. para
diferenciarlos de los de las bases nitrogenadas.
Fig. 3: Estructura de las pentosas constituyentes de ARN y ADN, respectivamente

Bioquímica General y Bucal - FOUBA
3
Nucleósido: Se forma al unirse una base nitrogenada con una pentosa mediante un enlace N-
glicosídico de configuración beta (Fig. 4), ya que conecta al átomo de C 1’ de la pentosa con el N-1 de
la base pirimidínica o con el N-9 de la base púrica. Los nucleósidos se nombran agregando el sufijo –
idina al nombre de la base pirimidínica que lo compone (citidina, timidina y uridina) y –osina si la base
es púrica (adenosina y guanosina).
Nucleótido:
Se forma por la unión de un nucleósido a través del oxhidrilo del C5’ de la pentosa con
el ácido fosfórico mediante un enlace
éster. Un ejemplo es el AMP o adenosin-5’-monofosfato (en
general: NMP= nucleósido-5’-monofosfato) ya que se nombra por el nucleósido que lo compone,
seguido de la posición numérica y cantidad de fosfatos unidos a la pentosa (Fig.4). Este primer fosfato
se denomina alfa. Los grupos fosfato adicionales se unen al fosfato de un NMP a través de un enlace
anhidrido (cuya energía de hidrólisis es alta siendo útil para realizar trabajo biológico) y se denominan
beta y gama, respectivamente. Siguiendo con el ejemplo se forman ADP o adenosin-5’-difosfato y ATP
o adenosin-5’-trifosfato (Fig. 5). Para diferenciar a los desoxirribonucleótidos en forma abreviada
usaremos una “d” antes de la sigla correspondiente (dAMP, dADP, dATP, dCMP, dCDP, dCTP, etc.). El
ATP es la “moneda energética” de la célula, el GTP es la mayor fuente de energía para la síntesis
proteica, el CTP es un metabolito esencial para la síntesis de fosfolípidos y el UTP forma intermediarios
activados con azúcares que sirven como sustratos en la biosíntesis de carbohidratos complejos y de
polisacáridos. Además los NTPs y los dNTPs son sustratos para la síntesis de los ácidos nucleicos.
Fig 4: Estructura de nucleósidos y nucleótidos

Bioquímica General y Bucal - FOUBA
4
Existen nucleósidos monofosfato en los cuales el ácido
fosfórico esterifico a dos de los oxhidrilos de la pentosa,
quedando formado un compuesto cíclico que resulta de
gran importancia como segundo mensajero regulador del
metabolismo celular. Los más importantes son el AMPc
(3’,5’-adenosin-monofosfato cíclico) y GMPc (3’,5’-
guanosin-monofosfato cíclico).
Fig 5: Estructura de nucleótidos mono, di y trifosfatos
Otras moléculas de tipo nucleotídico son las coenzimas como el FAD (flavinadenindinucleótido), NAD
(nicotinamidadeninadinucleótido), NADP (NAD fosfato) y la coenzima A (CoA), que son fundamentales
en el metabolismo celular (Fig.6).
Fig 6: Estructura de moléculas de tipo nucleotídico como el FAD
ADN (ÁCIDO DESOXIRRIBONUCLEICO)
El ADN aislado de células eucariotas consiste en dos cadenas polinucleotídicas unidas mediante uniones
puente de hidrógeno intercatenarias para formar una doble hélice complementaria de ADN (Fig 7).
Una cadena polinucleotídica es un polímero lineal de nucleótidos que están unidos covalentemente,
denominándose al enlace fosfodiéster 3’5’ ya que se formó por deshidratación entre el OH 3’ de un
nucleótido y el fosfato 5’ de otro nucleótido.
En 1953, James Watson y Francis Crick dedujeron la estructura tridimensional del ADN y propusieron
un modelo estructural que ha mostrado ser esencialmente correcto. Las características más
sobresalientes son:
1.
Hay dos cadenas helicoidales de polinucleótidos enrolladas (sentido horario o dextrógira) a lo
largo de un eje común. Las cadenas corren en direcciones opuestas, es decir, son
antiparalelas (una va
de 5’->3’ y la otra de 3’->5’).
2.
Las bases púricas y pirimidínicas están en el interior de la hélice(es la parte variable), mientras
que las unidades de fosfato y desoxirribosa están en el exterior (es la parte constante). Los planos que
contienen las bases son perpendiculares al eje de la hélice. Los planos que contienen los azúcares están
formando casi ángulos rectos con los de las bases.

Bioquímica General y Bucal - FOUBA
5
3.
Las dos cadenas permanecen unidas por
puentes de hidrógeno entre los pares de bases, siendo
el apareamiento de bases altamente específico (Chargaff). Siempre se enfrentan una base púrica con
una pirimidínica ya que ajustan perfectamente en el espacio disponible en el interior de la hélice. El
espacio sería insuficiente para dos purinas y demasiado grande para dos pirimidinas. Son
complementarias: adenina con timina y guanina con citosina ya que los átomos de hidrógeno de las
bases tienen posiciones espaciales muy definidas que restringen la formación de puentes de H de otro
modo.
4.
El diámetro de la hélice es de 20 Å (2nm). Las bases adyacentes están separadas 3,4 Å a lo largo
del eje de la hélice y la estructura helicoidal se repite en cada cadena después de 10 bases (a intervalos
de 34 Å por vuelta), describiendo un surco mayor y uno menor (Figura 8).
5.
La
secuencia de bases en una cadena mantiene una relación complementaria a la secuencia de
bases de la otra cadena y es la que transporta la información genética (Figuras 7 y 8).
Fig 7. Complementariedad y estabilización de las bases
Fig.8: Estructura tridimensional de la doble hélice de
ADN
Estabilidad de la doble hélice de ADN:
1.
Puentes de hidrógeno: a) Internos: entre las cadenas de ADN por complementariedad de bases
púricas y pirimidínicas (2 entre pares A-T y 3 entre pares G-C). b) Externos: entre los grupos polares del
esqueleto azúcar-fosfato y moléculas de agua circundantes.
2.
Interacciones Electrostáticas: Los grupos fosfato, cargados negativamente, están situados en la
superficie exterior de la doble hélice de modo que la repulsión electrostática entre ellos resulta mínima
y así se encuentran libres para interactuar electrostáticamente con cationes disueltos como el Mg
2+
.
3.
Interacciones Hidrofóbicas y Fuerzas de Van der Waals: En el corazón de la hélice entre las
bases estas fuerzas contribuyen a la estabilización energética general.
Propiedades de la duplicación del ADN:
La duplicación ocurre siempre en la fase S del ciclo celular de las células eucariotas
(https://docs.google.com/document/d/1m03zU_YlhCnCu_mB_vsvXiP7vvalZjrHxoKfeKrvcDg/edit?usp=

Bioquímica General y Bucal - FOUBA
6
sharing) y es semiconservativa pues cada molécula de ADN hija estará formada por una cadena
parental y una cadena nueva, recién sintetizada, por ello es esencial que las cadenas se separen y sirvan
de guía o molde para la formación de una nueva cadena. La duplicación comienza en un punto fijo en
el cual la secuencia de bases actúa como señal para el reconocimiento de las enzimas y factores que
participarán del proceso.
En células eucariotas existen numerosos orígenes de replicación a lo largo de todo el genoma. Durante
esta fase, los orígenes de replicación son activados en unidades de replicación o replicones hasta que
todo el ADN está replicado. En cada origen la duplicación es
bidireccional ya que se forman dos
horquillas de replicación que avanzan en direcciones opuestas, provocando la formación de burbujas
de replicación, que se van fusionando y desaparecen al finalizar la duplicación (figura 9).
Figura 9: Replicación bidireccional del ADN
Mecanismo:
La rapidez y exactitud de las enzimas de replicación se ha logrado mediante su asociación en un
verdadero complejo multienzimático de replicación. Analizaremos la función de cada una por orden de
aparición en el proceso y luego al complejo integrado como un todo.
Como mencionamos anteriormente, al comenzar el proceso las cadenas de ADN deben separarse en un
sitio preciso: en el origen de replicación (posee una secuencia de bases definida). Allí comienza la
formación de la horquilla de replicación que se logra por la acción combinada de las primeras tres
enzimas
1.
Proteína Iniciadora: Se une al origen de replicación, señalándolo.
2.
Helicasa: Se une a la proteína de iniciación y cataliza la ruptura de los puentes de H entre las
bases de las cadenas de ADN, abriendo la hélice. Utiliza ATP en el proceso.
3.
Primasa: Se une a la helicasa formando un complejo enzimático llamado primosoma y sintetiza
pequeños fragmentos de ARN llamados cebadores (“primers” en inglés) en sentido 5’->3’.
4.
ADN girasa (también llamada topoisomerasa II): Cataliza la relajación del superenrollamiento
de la doble cadena de ADN cortando por delante de la horquilla una cadena, desenrollando y volviendo
a unir. Utiliza ATP en el proceso.
5.
Proteínas desestabilizadoras de la hélice: También se conocen con el nombre de proteínas de
unión a ADN simple cadena (SSB), estabilizan las hebras e impiden la reasociación.
6.
ADN Polimerasa: Para comenzar a unir nucleótidos requiere de un OH 3’ que es el extremo del
cebador que sintetizó la primasa. Avanza a lo largo de las horquillas de replicación siguiendo al
primosoma, polimerizando nucleótidos complementarios a la cadena parental que está copiando. Los
sustratos de esta enzima son los desoxirribonucleósidos trifosfato (dATP, dGTP, dCTP y dTTP) que llevan
la energía necesaria para formar cada enlace fosfodiéster en sus dos enlaces anhídrido fosfórico y al

Bioquímica General y Bucal - FOUBA
7
liberarse en PPi permiten la unión de cada nucleótido (dNMP. Así la polimerización ocurre en sentido
5’->3’. El sentido de síntesis 5’->3’ se debe a la necesidad de alta fidelidad de copiado del material
genético que es vital para la supervivencia y proliferación celular. La polimerasa requiere de un extremo
OH3’ libre correctamente dispuesto en la cadena en formación (apareamiento complementario con la
cadena de ADN molde) y por eso tiene la capacidad de verificación de lectura. Si no fue correcto, extrae
el dNMP incorrecto con su actividad exonucleasa 3->5’ e incorpora uno correcto usando un nuevo dNTP
como sustrato.
El sentido obligado de síntesis 5’->3’ y la orientación antiparalela de las dos cadenas que serán
copiadas plantea un problema: la polimerasa sigue al primosoma mientras éste avanza abriendo la
horquilla de replicación de manera que una hebra queda expuesta en sentido 5’->3’ y la otra en sentido
3’->5’. La hebra 3’
->
5’ del ADN molde recibe el nombre de cadena conductora patrón o líder ya que
será copiada sin dificultad por la polimerasa que sintetiza la cadena conductora hija en forma continua
a partir del ARN cebador. En cambio, la hebra 5’->3’ del ADN molde se denomina cadena rezagada
patrón o retrasada pues será copiada con retraso por la polimerasa que sintetiza la cadena rezagada
hija en forma discontinua (en fragmentos). Esta síntesis es más lenta debido a que la polimerasa debe
esperar a que la cadena rezagada patrón quede expuesta (desapareada de la conductora patrón).
La polimerasa requiere de tantos ARN´s cebadores como fragmentos de ADN necesite sintetizar, todos
ellos serán sintetizados por la ARN primasa. Estas cortas cadenas de
ARN cebador (aproximadamente
10 nucleótidos) + ADN (aproximadamente 200 nucleótidos) se las conoce con el nombre de fragmentos
de Okazaki
De esta manera, se requieren dos ADN polimerasas –una para sintetizar la cadena continua y la otra
para la discontinua– que forman parte del complejo de replicación. En cambio, sólo hace falta un
primosoma (helicasa + ARN primasa) en cada complejo, pues primero la helicasa abre la hélice y la
primasa sintetiza el cebador de la cadena continua y luego se ocupa de sintetizar los cebadores de la
discontinua a medida que la helicasa sigue abriendo la hélice.
Las polimerasas de un complejo se frenan cuando se encuentran con otro complejo de replicación
correspondiente a la burbuja de replicación vecina. Aclaremos que en cada burbuja de replicación habrá
dos complejos actuando simultáneamente desde el origen de replicación alejándose en sentidos
opuestos (replicación bidireccional), provocando cada uno una horquilla. El desplazamiento de las
horquillas provoca superenrollamientos de la hélice de ADN entre las burbujas, que en exceso frenarían
el proceso de duplicación Para evitarlos existen enzimas llamadas
Para completar el proceso de duplicación deben reemplazarse todos los ARN cebadores por ADN esto
requiere de:
1.
Enzimas que degradan los ARN cebadores: Eliminan los cebadores para que la ADN polimerasa
complete los espacios vacíos con ADN complementario a esa región del molde.
2.
ADN Ligasa: une ADN que encuentra discontinuo, mediante un enlace fosfodiéster, formando
una cadena continua de ADN.
Así quedan formadas dos hélices de ADN con la misma información que la hélice que les dio origen.

Bioquímica General y Bucal - FOUBA
8
Figura 10: complejo multienzimático de replicación del ADN
https://www.youtube.com/watch?v=3gHskKXtRnA
PROCESO DE TRANSCRIPCIÓN O SÍNTESIS DE ARN EN CÉLULAS DE
EUCARIOTAS:
ARN (ácido ribonucleico): La estructura química difiere de la del ADN en
algunos aspectos (Fig. 11):
1.
La base timina presente en el ADN, es sustituida por uracilo en el
ARN. El uracilo posee un átomo de H, en vez de un grupo metilo (-CH
3
),
unido a uno de sus carbonos.
2.
El azúcar presente en el ARN es la ribosa, que posee un grupo OH
en su carbono 2’, en vez de un H, como la desoxirribosa del ADN.
3.
Generalmente se lo encuentra como una
cadena simple de ARN
con posibles regiones de apareamiento de secuencias de bases
complementarias por plegamiento.
Figura 11: estructura química del ARN
En células de eucariotas encontramos 11% de ARN en el núcleo, 15% en las mitocondrias, 50% en los
ribosomas y 24% en el citosol. Los distintos tipos de ARN se diferencian por su longitud, estructura y
funciones. Ellos son:
1.
ARNnh (nuclear heterogéneo): Constituyen el producto primario de la polimerasa II y
también se los puede llamar transcriptos primarios. Su maduración post- transcripcional dará lugar a
los ARNm (sólo un 20% completa este proceso).
2.
ARNm (mensajero): Son la matriz intermediaria o molécula puente para transformar la
información genética contenida en la secuencia de bases del ADN en la secuencia de aminoácidos de
las proteínas. Su tamaño puede variar desde 75 a 3000 nucleótidos

Bioquímica General y Bucal - FOUBA
9
3.
ARNt (de transferencia): Son los transportadores de aminoácidos durante el proceso de síntesis
proteica. Cada aminoácido posee por lo menos un ARNt específico, destinado a insertarlo en la cadena
polipeptídica en crecimiento. Su longitud aproximada es de 73 a 96 nucleótidos y generalmente
presentan bases metiladas.
4.
ARNr (ribosomal): Pertenecen a las estructuras ribonucleoproteicas (formadas por ARN (65%) y
proteínas (35%)) llamadas ribosomas, que constituyen el sitio donde se lleva a cabo la síntesis de
proteínas. Su tamaño varía desde 120 a 2900 nucleótidos y presentan bases metiladas. Los ribosomas
poseen una subunidad grande (3 ARN + 40 proteínas) y una pequeña (1 ARN + 30 proteínas).
5.
ARNnp (nucleares pequeños) o RNAsn (“small nuclear” en inglés): Se encuentran en el núcleo
de células eucariotas formando parte de complejos estables con proteínas específicas llamadas
ribonucleoproteínas nucleares pequeñas (RNPnp) o RNPsn. Su longitud es de 100 a 200 nucleótidos,
algunos metilados. Participan tanto en el proceso de corte del transcrito primario por el sitio donde se
añadirá la cola de poli-A como en la localización y eliminación de los intrones durante la maduración
de los ARNm (conceptos que se aclaran más adelante).
Mecanismo de Transcripción:
La síntesis de ARN es un proceso muy selectivo. En la mayor parte de las células sólo se copian las
secuencias de ARN funcionales (mensajeros o estructurales) que resultan alrededor del 1% de las
secuencias de nucleótidos del ADN.
En eucariotas existen tres enzimas diferentes que catalizan la polimerización de nucleótidos que darán
lugar a los distintos tipos de ARN:
ARN polimerasa I: para ARNr correspondiente a la subunidad grande.
ARN polimerasa II: para ARNm.
ARN polimerasa III: para ARNr correspondiente a la subunidad pequeña y ARNt.
Cada una de ellas requiere de un factor proteico específico, llamado factor de transcripción (FT) I, II o
III, respectivamente, unido al ADN señalando la región donde va a comenzar la transcripción. Esta zona
del ADN, llamada promotor, determina cuál de las cadenas de ADN será el molde y contiene secuencias
específicas donde se unirá el FT correspondiente y también el sitio de iniciación de la transcripción.
Síntesis de ARNm:
El TFII también llamado factor TATA - ya que se une a secuencias ricas en A-T denominadas caja TATA,
situadas en el ADN entre 20 y 30 nucleótidos por delante del lugar de inicio de la transcripción - se une
al promotor activándolo (haciéndolo funcional). Entonces, comienza la transcripción cuando la enzima
polimerasa II (ARN pol II) se une al promotor + FTII y desenrolla parte de la doble hélice (formando un
complejo abierto), dejando expuestas dos cadenas sencillas de ADN, una de las cuales se transcribirá
(sólo una será el molde para la síntesis del ARNm). (Fig 12).
Fig 12: Iniciación de la transcripción. Formación del complejo de preiniciación

Bioquímica General y Bucal - FOUBA
10
A medida que la polimerasa recorre el ADN, nucleótidos de ARN con bases complementarias a las que
porta el ADN molde se van añadiendo uno a uno a la cadena de ARN que se está sintetizando. Cada NTP
que se va a incorporar lleva un grupo trifosfato en la posición 5’ que durante la reacción de síntesis
libera PPi, quedando unido el P 5’ de dicho nucleótido con el grupo OH 3’ del nucleótido situado al final
de la cadena mediante un enlace fosfodiéster (O-P-O). La molécula de ARN crece en dirección 5’->3’ de
la misma forma que lo hace el ADN durante su replicación. Entonces, el ADN molde será la cadena con
sentido 3’->5’ (Fig 13).
Fig. 13: Iniciación de la transcripción. Alargamiento de los primeros 10 nucleótidos
Cuando el transcrito mide aproximadamente 30 nucleótidos, se añade una “caperuza” o CAP (en inglés)
a su extremo 5’. Esta caperuza es una guanosina metilada (7mG) y con un grupo trifosfato que se une
en forma poco habitual 5’ -> 5’. La polimerasa continúa trabajando hasta que transcribe la secuencia
de bases AAUAAA que actúa de señal de terminación ya que unos 20 nucleótidos curso abajo se corta
el transcrito, reacción en la que está involucrada una ribonucleoproteína nuclear pequeña (RNPnp) o
RNPsn que contiene un “ARN U1”, rico en la base uracilo (Fig. 14).
Fig. 14: Elongación y terminación de la transcripción.
Una enzima polimerasa especial agrega una “cola” de 150 a 200 nucleótidos de adenina
(poli-A) al
extremo 3’ completando el transcrito primario de ARN (Figura 14). La polimerasa continúa
transcribiendo de forma inútil (ya que el ARN formado no tiene CAP en 5’ y por lo tanto es degradado
rápidamente) hasta que se desprende del ADN molde en lugares de terminación posteriores. El ADN
queda con su forma original de doble hélice.
https://www.youtube.com/watch?v=6rvlyYpaEaQ
Uno de los avances más espectaculares en el desarrollo de la biología molecular ha sido el
descubrimiento de que muchos genes eucarióticos están fragmentados y que el transcrito primario de

Bioquímica General y Bucal - FOUBA
11
ARN debe sufrir un proceso de cortes y empalmes, denominado maduración del ARNm o splicing. El
ARNm no es una copia “literal” del ADN, sino que en su maduración se elimina una cantidad importante
de material. Las secuencias del transcrito primario que codifican secuencias proteicas se llaman
exones,
mientras que las que no codifican, intrones. Entonces, los exones estarán presentes en el ARNm
maduro y los intrones serán eliminados. En el proceso colaboran RNPsn que tienen ARN U1 (entre otros)
que es capaz de aparearse con una secuencia que existe al principio de cada intrón en la que están
presentes las bases GU. Entonces las RNPsn asociadas al ARN transcrito primario forman un complejo
llamado
spliceosoma (su ensamble requiere de ATP) que facilitan el acercamiento de los extremos de
los exones y su empalme, hecho que produce la formación de los lazos de intrones y su liberación,
siendo rápidamente degradados en el núcleo (Fig. 15).
Fig. 15 Maduración del transcripto primario de ARNm: Splicing.
El proceso de Splicing descripto puede ser de dos tipos:
Constitutivo: Cuando cada intrón es removido y todos los exones son incorporados al ARNm
maduro, de modo que se produce una única forma de ARNm maduro a partir de un transcrito primario.
Alternativo: Cuando se producen múltiples formas de ARNm maduros a partir de un transcrito
primario, debido al empalme diferencial de algunos de los exones.
Bioquímica General y Bucal - FOUBA
12
REGULACIÓN DE LA EXPRESIÓN GÉNICA:
Cada región del ADN que produce una molécula de ARN funcional constituye un gen. Los genes de un
cromosoma de un eucarionte superior pueden contener desde 100.000 a 2.000.000 pares de
nucleótidos de ADN. Sin embargo, entre 300 y 3000 nucleótidos se necesitan para codificar una proteína
de tamaño entre 100 y 1000 aminoácidos. Por lo tanto, existen largas porciones de ADN no codificantes
(intrones) y segmentos cortos de zonas codificantes (exones), además de las secuencias regulatorias
que controlan la transcripción del gen.
El mecanismo predominante de control es la transcripción alternativa de genes (decisión de cada célula
de qué genes se van a transcribir).
Conocida la naturaleza fragmentada de los genes, el proceso de maduración del ARN, también
interviene en la regulación de la expresión génica:
Dentro de una célula, los niveles de proteínas pueden venir determinados por la cantidad de ARNm
que llega a madurar.
Además, un gen puede determinar más de una proteína (proteínas isoformas), y la decisión de cuál
fabricar en un determinado momento o en una célula dada, puede depender del tipo de maduración
que se siga (alternativa).
Durante la transcripción: a) un transcrito primario con dos o más sitios donde colocar la secuencia
poli-A puede cortarse por cualquiera de esos puntos, resultando en cada caso un ARNm con un
extremo 3’ distinto y por tanto una proteína diferente. B) La utilización de diferentes promotores
puede generar ARNm con diferentes extremos 5’, y así también proteínas diferentes.
En todos los transcritos primarios de ARN (mensajero, ribosomales y de transferencia) quedan las
secuencias codificantes (exones) y no codificantes (intrones), de modo que todos sufren el proceso de
maduración o splicing. Lo que
diferencia a los ARNr y ARNt del ARNm son los extremos CAP 5’ y poli-
A 3’, presentes sólo en el ARNm. Cuando los ARN están maduros, son transportados al citoplasma a
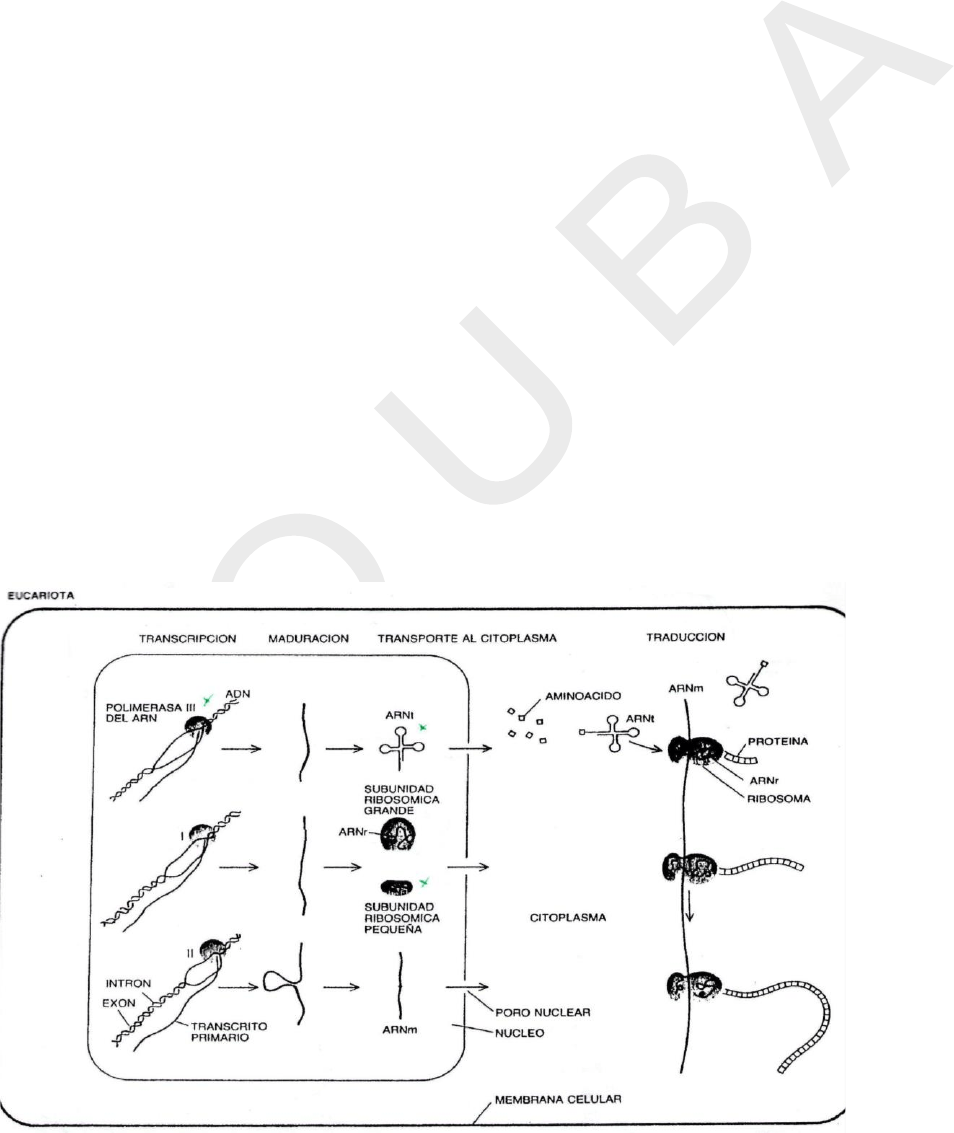
través de los poros nucleares para realizar la síntesis de proteínas (Fig. 16).
Figura 16:
Transcripción,
maduración,
transporte al
citoplasma de
todos los ARN
(ARNm, ARNr
y ARNt) en
una célula
eucariota.

Bioquímica General y Bucal - FOUBA
13
PROCESO DE TRADUCCIÓN O SÍNTESIS DE PROTEÍNAS EN CÉLULAS DE EUCARIOTAS:
Código Genético (Tabla I):
Son las reglas a través de las cuales se traduce la secuencia nucleotídica en secuencia de aminoácidos
de una proteína que fueron descifradas a principios de los años 60. Se demostró que la secuencia de
nucleótidos de la molécula de ARNm era leída en orden consecutivo, en grupos de tres. Cada triplete
de nucleótidos, denominado
codón, determina un aminoácido. El código genético es universal, ya que
los codones tienen el mismo significado en todos los organismos vivos, degenerado debido a que la
mayoría de los aminoácidos son especificados por más de un codón. Esto se debe a que el ARNm es
un polímero lineal de cuatro nucleótidos diferentes y por lo tanto, existen 4
3
= 64 tripletes de codón
posibles, mientras que en las proteínas habitualmente se encuentran sólo 20 aminoácidos diferentes y
no es ambiguo ya que un codón sólo indica un aminoácido.
Tabla I: Código genético.
Bioquímica General y Bucal - FOUBA
14
Moléculas necesarias para la síntesis de proteínas:
1)
ARNt :
Son moléculas adaptadoras ya que se
unen por uno de sus extremos (llamado
anticodón) a un codón específico de la
molécula de ARNm y por el otro al
aminoácido específico para ese codón.
La estructura tridimensional se asemeja
a una hoja de trébol, que
posteriormente se pliega adoptando la
forma de L (Figura 17).
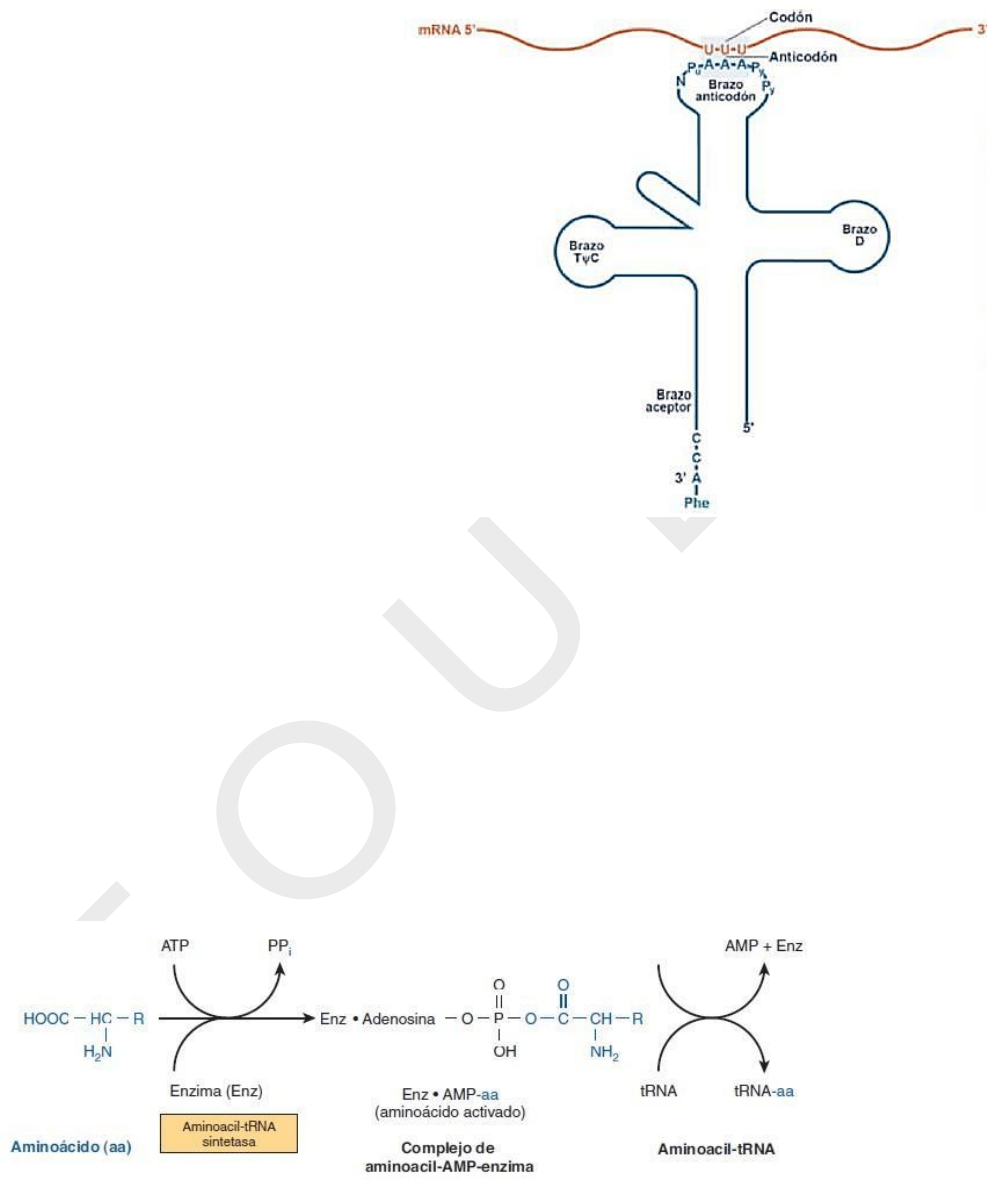
Fig. 17: Estructura
tridimensional en forma de trébol del ARNt
El
aminoácido se une a un extremo de la L activándose, a través de un enlace covalente de tipo éster.
La enzima responsable de unir cada aminoácido a su grupo de moléculas de ARNt apropiadas se
denomina
aminoacil-ARNt sintetasa. Existe una sintetasa diferente para cada aminoácido. Por
ejemplo, para la Glicina (Gly) hay 4 codones posibles (tabla I) (ellos son: GGU, GGC, GGA y GGG), y por
lo tanto habrá 4 ARNt con sus 4 anticodones correspondientes que serán cargados por una sintetasa
específica para Gly para formar los ARNt
gly
. La reacción de activación se realiza en dos etapas (Fig.18):
1)
Se forma un aminoácido adenilado por unión de su grupo alfa-carboxilo con el fosfato de un
AMP. Para ello se requiere de la hidrólisis de los dos enlaces de alta energía del ATP, liberándose PPi.
2)
El grupo carboxilo unido al AMP es transferido a un grupo OH 3’ del azúcar del extremo del ARNt,
formándose un
aminoacil-ARNt con una unión éster, rica en energía, en el extremo carboxílico del
aminoácido que será necesaria para formar el enlace peptídico.
Fig. 18: Aminoacil-ARNt sintetasa. Activación de los aminoácidos.
En el otro extremo se ubica el
anticodón – secuencia del ARNt donde se une el codón correspondiente
del ARNm. Entonces, es la molécula de ARNt, y no el aminoácido que ésta lleva unido, la que determina

Bioquímica General y Bucal - FOUBA
15
el lugar en el que será incorporado el aminoácido durante la síntesis proteica. Por eso es esencial la
precisión de la aminoacil-ARNt sintetasa en la elección del aminoácido correcto que debe unir.
2)
Ribosomas:
Son la maquinaria catalítica que guía la síntesis de proteínas permitiendo la interacción de todos los
componentes que intervienen. Cada uno de ellos está compuesto por dos subunidades.
La
subunidad mayor posee tres sitios de unión para moléculas de ARN: uno para ARNm y dos para
ARNt: 1) el lugar de
unión del peptidil-ARNt o sitio P que acoge al ARNt que lleva unido el extremo de
la cadena polipeptídica en crecimiento y 2) el lugar de unión del aminoacil-ARNt o sitio A que acoge a
la molécula de ARNt entrante cargada con un aminoácido (Figura 19).
Para que una molécula de ARNt se una fuertemente a cualquiera de
estos dos lugares, es necesario que su anticodón forme los pares de
bases complementarios al codón del ARNm que está unido al
ribosoma. Los sitios A y P están tan cerca el uno del otro que las dos
moléculas de ARNt se ven forzadas a aparearse con codones
adyacentes de la molécula de ARNm. Así se asegura de no saltear
ningún nucleótido del ARNm que está traduciendo.
La subunidad grande cataliza la formación de los enlaces peptídicos, ya
que posee la actividad de peptidil-transferasa.
Fig. 19: Ribosoma con los lugares de unión de todos los componentes que intervienen
.
Mecanismo:
El proceso de síntesis proteica se divide en tres etapas:
Iniciación (Figura 20):
a)
Primero se unen factores de iniciación a la subunidad pequeña del ribosoma, que de este modo
puede unirse al ARNm identificando su extremo 5’ por la caperuza que éste lleva.
b)
Luego un
aminoacil-ARNt iniciador que siempre es Metionil-ARNt (en eucariotas) unido a un
factor de iniciación 2 de eucariotas (eIF-2) se carga a la subunidad pequeña del ribosoma que ya está
unida al ARNm. Así se desplazan en conjunto en sentido 5’->3’ hasta encontrar el primer codón AUG
en el ARNm
. Esto garantiza un único marco de lectura
para un determinado ARNm ya que normalmente
existen varias secuencias AUG y sólo una de ellas es
reconocida como codón de iniciación. Entonces, queda
el
Metionil-ARNt + eIF-2 en el sitio P.
c)
Una vez ubicado el codón de iniciación los
factores de iniciación se separan de la subunidad
pequeña permitiendo que a ella se le una la subunidad
grande.
Así finaliza la etapa de iniciación, con la
maquinaria ensamblada en el sitio correcto y
comienza la segunda etapa.
Fig 20: Iniciación de la traducción (síntesis de proteínas)
Este documento contiene más páginas...
Descargar Completo
Material de estudio Módulo 6. Acidos nucleicos.pdf
 Estamos procesando este archivo...
Estamos procesando este archivo...
 Lamentablemente la previsualización de este archivo no está disponible. De todas maneras puedes descargarlo y ver si te es útil.
Lamentablemente la previsualización de este archivo no está disponible. De todas maneras puedes descargarlo y ver si te es útil.
Descargar
 Estamos procesando este archivo...
Estamos procesando este archivo...
 Lamentablemente la previsualización de este archivo no está disponible. De todas maneras puedes descargarlo y ver si te es útil.
Lamentablemente la previsualización de este archivo no está disponible. De todas maneras puedes descargarlo y ver si te es útil.