1
I.S.U Instituto Superior Universitario
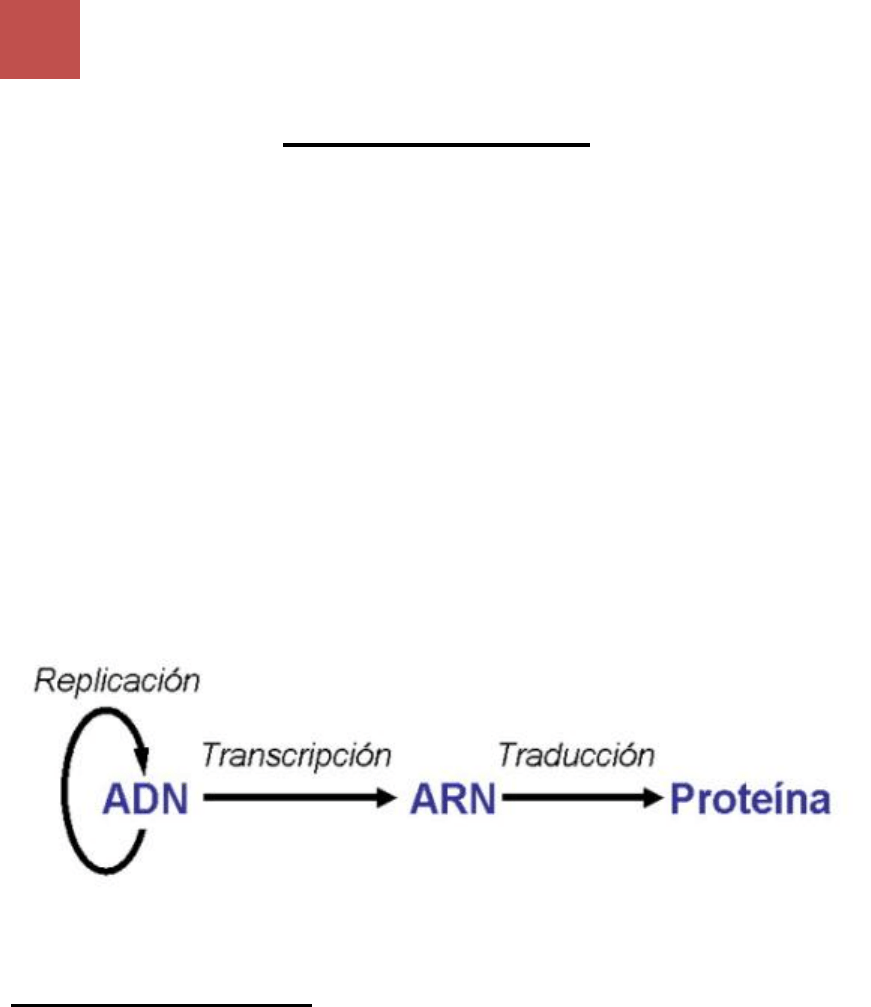
BIOSINTESIS DE ADN
El genoma de cada individuo está contenido en moléculas de ADN de sus cromosomas y
mitocondrias, también en los cloroplastos de las células vegetales.
En ellas se encuentran las unidades de información llamadas GENES. Los genes son porciones
de ADN que dirigen la síntesis de distintas especies de ARN funcionales en las células:
ribosomal, de transferencia, mensajero, entre otros.
El proceso de duplicación o REPLICACION del ADN, se lleva a cabo durante la división celular.
Para expresarse, la información que el ADN contiene, debe ser transferida a moléculas de ARN
por un proceso denominado TRANSCRIPCION.
La secuencia de bases del ARNm indica el orden en el cual se van a ensamblar los aminoácidos
de las proteínas. La síntesis de cadenas polipeptídicas requiere de la TRADUCCION del mensaje
cifrado en ARNm.
REPLICACION DEL ADN
Es un mecanismo mediante el cual todas las células somáticas originadas por divisiones
sucesivas de una única célula poseen igual cantidad de ADN con la misma estructura primaria.
La replicación del ADN es SEMICONSERVADORA, esto se debe a que la cadena de ADN sirve
como molde o guía para la síntesis de una nueva hebra polinucleótida complementaria.
Entonces cada célula hija tendrá una cadena nueva y la otra que procede de la progenitora. La
replicación tiene lugar en la fase S del ciclo celular.

2
I.S.U Instituto Superior Universitario
CICLO CELULAR
EXISTEN MÚLTIPLES ENZIMAS Y FACTORES QUE INTERVIENEN
EN EL PROCESO DE REPLICACIÓN DE LAS CÉLULAS EUCARIOTAS
Y PROCARIOTAS SIENDO MÁS COMPLEJO EL PRIMERO.
PASO INICIAL:
el primer paso de la replicación es la separación de las dos cadenas, este
proceso es indispensable para que cada uno sirva de molde o guía. La replicación comienza en
un punto fijo, el SITIO DE ORIGEN U ORI que posee secuencias de bases específicas que sirven
de señales de reconocimiento para el complejo de enzimas y factores que inicie la replicación
(generalmente posee secuencias ricas en A-T, más fáciles de separar). Este sitio de origen es
único en bacterias, ADN virales y mitocondrias. Por el contrario la separación en las células
eucariotas se inicia simultáneamente en todos los cromosomas y en muchos puntos de la
molécula de ADN de cada uno de ellos, originando BURBUJAS O BUBONES las cuales reciben el
nombre de REPLICONES. En cada burbuja o zona de separación se producen dos sitios de
separación, cada uno se llama HORQUILLA DE SEPARACION. Dichas oquillas avanzan en
sentidos opuestos alejándose del sitio de origen.
Esquema de la replicación del ADN:
A. Molécula original (doble hélice).
B. Se inicia la separación y replicación simultánea de ambas hélices de ADN.
C. Se ha completado la replicación y se han formado dos dobles hélices de ADN
iguales a la original

3
I.S.U Instituto Superior Universitario
Las cadenas originales son la de los extremos y las nuevas las que se encuentran en el
interior.
En el lugar de origen de la replicación, señalado por secuencias específicas, se unen
múltiples subunidades de una proteína iniciadora. A continuación se forma un complejo de
varias enzimas entre ellas:
a) HELICASA: o enzima desarrollante, se une al ORI, cataliza la separación de las
cadenas, para esto necesita energía, dada por la hidrólisis del ATP.
b) TOPOISOMERASA: realiza cortes transitorios de la cadena de ADN, para evitar
zonas hipertorsionadas al separar las hebras. Hay dos tipos: I, la cual corta una de
las hebras de la doble hélice y alivia la tensión por rotación libre de la cadena,
seccionada sobre otra. II, también llamada girasa, secciona las dos hebras de ADN.
Ambas tienen capacidad para unir los cabos de las cadenas cortadas y restablecer
la continuidad de la doble hélice en estado relajado.
c) Existen proteínas específicas que mantienen las hebras separadas: a medida que
las hebras de ADN se separan, se unen a ellas proteínas fijadoras de ADN
monocetenario (SSB), estabilizan las hebras e impiden su reasociación. La
encargada de esta función en eucariotas es llamada proteína A de replicación
(RPA). La helicasa y las SSB avanzan a lo largo de la doble hélice, dejando atrás las
dos cadenas separadas, listas para la replicación.
d) ADN polimerasa: son enzimas que catalizan el ensamblaje de desoxirribonucleicos
para la formación de la nueva cadena. En bacterias existen tres tipos: I, II, III. En
eucariotes se conoce 5 tipos: α, β, γ, δ, ε. “DIFIEREN ENTRE SI EN SUS
PROPIEDADES Y FUNCIONES”. Sin embargo todas ellas comparten las siguientes
características:
1. Sus sustratos son los 4 dexorribonucleósidos trifosfato d ATP, dGTP, dCTP,
dTTP. Los cuales aportan materia prima para la síntesis y también de la
energía requerida.
2. Necesitan de una cadena molde.
3. Sólo pueden elongar una hebra preexistente llamada “cebador o primer”.
(cadena preexistente de ARN y es sintetizada por otra enzima llamada
primasa).
A
B
C
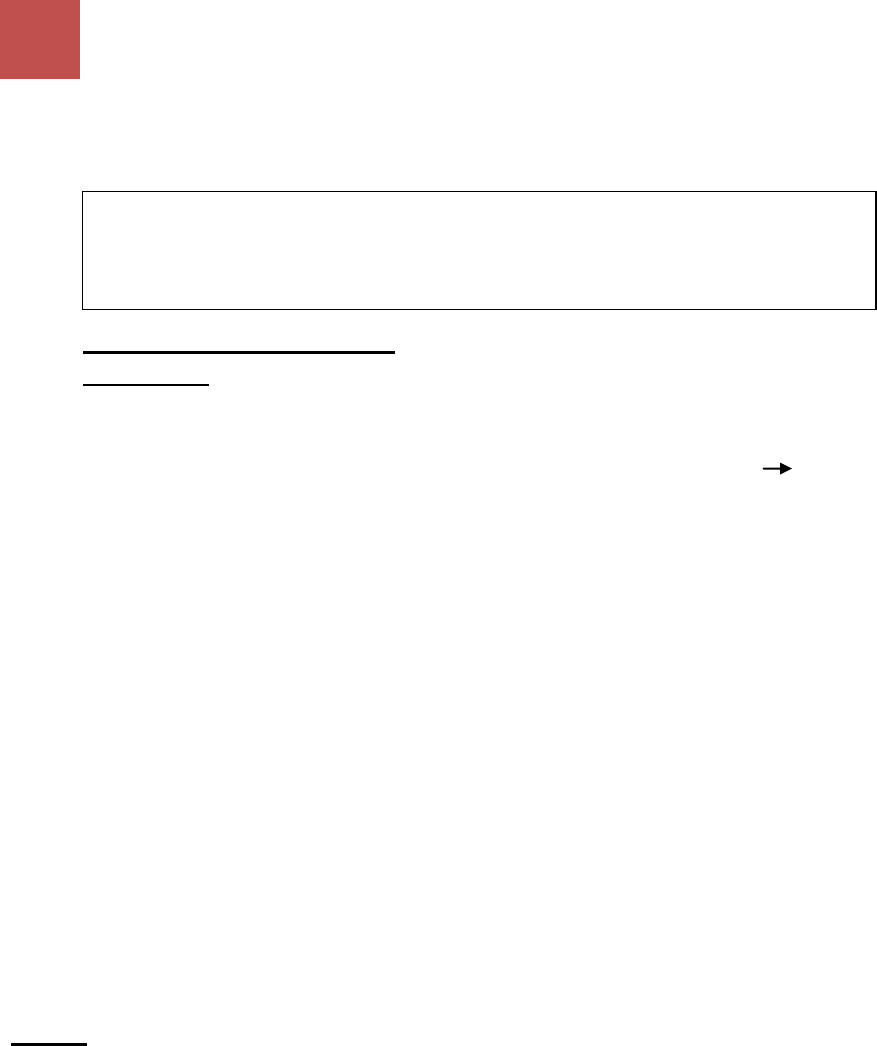
4
I.S.U Instituto Superior Universitario
4. Todas catalizan la unión de desoxirribunocleótidos para formar cadenas
desde el extremo 5´hacia 3´. La replicación en sí la lleva a cabo la ADN
polimerasa.
EL CONJUNTO DE TODAS LAS PROTEÍNAS EN LA DUPLICACIÓN DEL ADN ES UN
COMPLEJO MULTIENZIMÁTICO QUE FUNCIONA COMO UNA VERDADERA “MÁQUINA
DE REPLICACIÓN”, A LA CUAL SE LA LLAMA REPLISOMA.
Mecanismo de replicación:
Primer paso:
formación del primer o cebador o iniciador (trozos de ADN de unos 10
nucleótidos de extensión). Ésta síntesis es catalizada por la ARN-polimerasa
denominada PRIMASA, firmemente asociada a la ADN polimerasa α (pol α) de
eucariotes. La primasa incorpora ribonucleósidos trifosfato en sentido 5´ 3´, para
formar el segmento complementario del ADN molde. Posteriormente comienza a
actuar la pol α, donde se agregan 20 desoxirribonucleótidos. Cuando se ha formado un
trozo de 30 nucleótidos, el complejo polα-primasa se desprende y la elongación de la
cadena es continuada por la polimerasa δ. En cada incorporación de nucleótidos se
libera PPi, el proceso es irreversible, exergónico y el PPi inorgánico es hidrolizado de
inmediato.
Cuando se produce la separación de las 2 hebras, una tendrá la dirección 3´5´
apropiada para la lectura por la ADN-polimerasa. Sobre esta cadena, la síntesis de la
nueva hebra complementaria prosigue sin interrupción desde la formación del
iniciador ARN-ADN. A esta hebra se la llama LIDER, CONDUCTORA O ADELANTADORA.
En la otra cadena, la síntesis se realiza en segmentos, ya que el sentido es contrario al
progreso de la ADN-polimerasa. Se forman alrededor de 200 nucleótidos de extensión
designados FRAGMENTOS DE OKASAKI. La cadena que se forma en trozos recibe el
nombre de RETARDADA O REGAZADA. Posteriormente se eliminan los trozos del ARN
iniciador de cada fragmento de OKASAKI, y se sintetiza ADN para cubrir la brecha y los
extremos 3´5´ de segmentos adyacentes son unidos para obtener una cadena
continua.
Pasos
1) La primasa asociada a la polimerasa α, cataliza la síntesis del “primer”, luego la pol α
agrega 20 dexorribonucleósidos. De inmediato el factor de replicación C (RFC), el cual
es una proteína que se une al extremo 3´del segmento ARN-ADN, desplaza al complejo
primasa-pol α. Posteriormente carga la proteína, Antígeno Nuclear de las Células
Proliferantes (PCNA) sobre la doble hélice en formación.
2) El PCNA promueve el ingreso de la pol δ y el complejo polδ-PCNA se desliza sobre la
cadena molde en sentido de la síntesis.
3) Después se elimina el segmento del ARN iniciador, función llevada a cabo por
ribonucleasas designadas H
1
y FEN1. Esta eliminación es cubierta por ADN sintetizado
por la polδ.
4) Finalmente se establecen enlaces fosfodiester entre segmentos vecinos dado por ADN
ligasa para finalmente obtener una cadena continua.

5
I.S.U Instituto Superior Universitario

6
I.S.U Instituto Superior Universitario
Función de otras polimerasas:
ADN polimerasa : intervienen en la replicación del ADN mitocondrial.
ADN polimerasa : participa en procesos de reparación de ADN.
ADN polimerasa : función similar a la pol , también participa en la reparación del
ADN.
Reparación de ADN
El primer agente de control de calidad es la ADN polimerasa
, mientras inserta nucleótidos
complementarios a la hebra guía, realiza una tarea de detección de errores (Proof-reading).
Las polimerasa
, , tienen actividad exonucleasa 3´5´, que les permite eliminar la última
base incorporada sino está adecuadamente apareada.

7
I.S.U Instituto Superior Universitario
La polimerasa
, y tienen la capacidad de eliminar un trozo defectuoso y reemplazarlo por la
secuencia correcta.
Telomerasas
Cuando se ha completado el proceso de duplicación del ADN, frente a los extremos 3´de
ambas hebras molde queda un trozo de ARN iniciador. Éstos segmentos se eliminan por acción
de ARNasas, pero no pueden ser reemplazados por ADN, pues ADN polimerasas no funcionan
sin primer. El cabo 3´de cada guía no puede ser duplicado. El problema se resuelve por adición
a los extremos, de trozos de ADN que no contienen información. En humanos el ADN
telomérico “descartable” está constituido por cientos de repeticiones de una secuencia corta
de bases (TTAGGG). La inserción de los segmentos al extremo 3´del ADN telomérico
preexistentes es catalizado por TELOMERASAS, enzimas que contienen en su estructura un
trozo de ARN utilizado como molde para ensamblar las porciones con las cuales se elonga la
cadena de ADN.
Enzimas y factores comprometidos en la replicación de ADN en
eucariotes.
PROTEINAS
FUNCION
HELICASA (PROTEINA DESARROLANTE)
SEPARA ALS DOS HELICES POR RUPTURA DE
LOS PUENTES DE HIDRÓGENO QUE
MANTIENEN APAREADAS LAS BASES
TOPOISOMERASAS I Y II
PRODUCEN CORTES DE LAS CADENAS DE
DOBLE HELICE POR DELANTE DE LA
HORQUILLA DE REPLICACION, PARA ALIVIAR
LAS TENSIONES CREADAS POR EL
DESARROLLAMIENTO.
PROTEINA A DE REPLICAION (RPA)
SE FIJA A LAS HEBRAS SEPARADAS DE ADN
ORIGINAL E IMPIDE SU REASOCIACION.
PRIMASA (ASOCIADA A POLIMERASA)
CATALIZA LA SINTESIS DE UN TROZO DE ARN
INICIADOR O PRIMER, DE UNOS 10
NUCLEÓTIDOS.
ADN POLIMERASA
CATALIZA LA SINTESIS DE UN TROZO DE ADN
DE UNOS 20 NUCLEÓTIDOS A CONTINUACION
DEL ARN INICIADOR.
FACTOR C DE REPLICACIÓN (RFC)
DESPLAZA AL COMPLEJO POLIMERASA-
PRIMASA. FIJA PCNA
ANTIGENO NUCLEAR DE LAS CELULAS
PROLIFERANTES (PCNA)
ESTRUCTURA EN ANILLO QUE FIJA
POLIMERASA Y ASEGURA LA SINTESIS

8
I.S.U Instituto Superior Universitario
ININTERRUMPIDA.
ADN POLIMERASA
CATALIZA LA SINTESIS DE ADN A
CONTINUACION DEL INICIADOR PRODUCIDO
POR POLIMERASA-PRIMASA. TIENE
ACTIVIDAD EXONUCLEASA 3´5´.
ARNASA HI NUCLEASA FEN1
ELIMINA LOS TROZOS DE ARN INICIADOR.
ADN LIGASA
UNE MEDIANTE ENLACES FOSFODIESTER
3´5´LOS SEGMENTOS DE ADN.
ADN POLIMERASA
SINTESIS Y REPARACION DE ADN. TIENE
ACTIVIDAD EXONUCLEASA 3´5´.
ADN POLIMERASA
SINTETIZA ADN MITOCONDRIAL CIRCULAR.
TIENE ACTIVIDAD EXONUCLEASA 3´5´.
ADN POLIMERASA
ACTÚA EN SISTEMA DE REPARACION DE ADN.
CARACTERÍSTICAS DE LA REPLICACION DEL ADN.
ANABÓLICA: es un proceso de síntesis, es decir, endergónico.
BIDIRECCIONAL: la burbuja de replicación crece hacia ambos extremos.
SEMICONSERVADORA: cada molécula hija posee una cadena nueva y proveniente
de la madre, osea, cada hija conserva una cadena original.

9
I.S.U Instituto Superior Universitario
ADN PROCARIOTA
A diferencia del eucariota, no se encuentra enroscado sobre octámeros de histonas, por otro
lado el cromosoma bacteriano es único y circular. Esto provoca que el proceso de replicación
tenga algunas diferencias con respecto al de los eucariotas.
A partir del único ORI comienzan a separarse las cadenas, se forma la única burbuja de
replicación que crecerá sin detenerse hasta terminar de replicar el cromosoma en un punto
diametralmente opuesto al ORI. A medida que la burbuja avanza se forma un estructura que
tiene el aspecto de la letra griega Theta, lo que se conoce como forma –theta. El ADN
eucariota presenta varios orígenes de replicación, las burbujas que se van formando a partir de
cada uno de ellos lo hacen asincrónicamente.

10
I.S.U Instituto Superior Universitario
TRANSCRIPCIÓN
Este proceso consiste en la formación de moléculas de ARN a partir de una cadena molde
de ADN, por lo que también puede definirse como el pasaje de información del ADN al
ARN. Sobre las bases expuestas de una cadena de ADN se construye una cadena de ARN
respetando la complementariedad de bases y el antiparalelismo de las cadenas.
La transcripción, a diferencia de la replicación, es un proceso localizado ya que no ocurre
sobre todo el ADN, sino, que sólo atañe a determinados segmentos que portan la
información requerida: los GENES. Para poder transcribir a esos genes, primero hace falta
ubicarlos en todo el ADN, y luego establecer el punto exacto de inicio de la transcripción.
Existe una secuencia particular, de bases que determina ese punto de iniciación. Es el
PROMOTOR, el cual es reconocido por una enzima que es la ARN polimerasa-ADN
dependiente (ARNpol).
TODOS LOS ARN SE GENERAN EN EL NÚCLEO, UNA VEZ COMPLETADA LA SÍNTESIS, LAS
MOLÉCULAS DE ARN SE DIRIGEN AL SITIO EN EL CUAL CUMPLIRÁN SU FUNCIÓN.
Esta ARN polimerasa dependiente posee las siguientes características:
Tiene gran afinidad por ADN de doble cadena.
Reconoce secuencias de iniciación (promotores).
Separa las cadenas.

11
I.S.U Instituto Superior Universitario
Tiene actividad polimerasa 5´3´. Toma ribonucleótidos trifosfato (NTPs: ATP, GTP,
CTP y UTP), a los que les quita pirofosfato (PPi) y los incorpora monofosfatados
uniéndolos por enlace fosfodiester.
No requiere primer.
Separa del molde de ADN, la cadena de ARN que va elaborando.
Reconoce secuencias de terminación que le indica detener su actividad.
La actividad de la ARN pol puede ser regulada ya que existen secuencias a las que
pueden unirse proteínas reguladoras capaces de estimular o inhibir su actividad.
La ARN pol, reconoce al promotor y se une a él, una vez ubicada, separa las cadenas, creando
así la burbuja de transcripción a fin de poder leer la secuencia de bases de la cadena de ADN
que le sirve de molde. Note que la ARN pol sólo lee una de las cadenas de ADN, que es la única
sobre la cual puede construir el ARN, en sentido 5´3´a partir del promotor. A esta cadena de
ADN se la conoce como la cadena Sense, y a su complementaria, la que no es leída, se la
denomina Antisense.
La ARN pol, va incorporando ribonucleótidos, respetando la complementariedad de bases del
molde, uniéndolos unos con otros mediante enlaces fosfodiester.
Cuando el ARN que está siendo sintetizado alcanza una longitud considerable comienza a ser
despegado del ADN molde a partir del extremo 5´del ARN. Las bases del ADN molde, al soltarse
del ARN, quedan reexpuestas y vuelven a formar uniones puentes de hidrógeno con las bases
de la otra cadena de ADN (complementaria). Así se reconstituye el ADN doble cadena por
donde ya pasó la ARN pol, mientras la burbuja, manteniendo su tamaño, se desplaza junto con
la enzima. Note la diferencia entre esta burbuja y la de replicación, la cual crece
continuamente y de modo bidireccional.
LA BURBUJA DE TRANSCRIPCIÓN NO CRECE INDEFINIDAMENTE Y AVANZA EN UN SOLO
SENTIDO.

12
I.S.U Instituto Superior Universitario
De esta manera la ARN pol, va copiando hasta encontrar alguna secuencia que indique FIN. La
cadena de ARN sintetizada es separada por completo del molde.
El ARN sintetizado es antiparalelo y complementario a la cadena SENSE (molde) y presenta la
misma secuencia de bases que la ANTISENCE, con la diferencia en que uno hay
desoxiribonucleótidos y en el otro ribonucleótidos.
El ARN a su vez se puede dividir en tres tipos diferentes:
ARN MENSAJERO (ARNm): catalizado por la ARN polimerasa II.
ARN DE TRANSFERENCIA (ARNt): catalizado por la ARN polimerasa III.
ARN RIBOSOMAL (ARNr): catalizado por la ARN polimerasa I en el núcleo.
Todo lo explicado hasta aquí es común a todos los tipos de ARN, así como a todos los tipos
celulares (procariota y eucariota). Sin embargo, existen algunas diferencias. En las células
eucariotas al enzima que transcribe cada uno de ellos es una ARNpol, diferente y también es
distinto el sitio del núcleo en el que ocurre la transcripción. Mientras que todos los ARN se
sintetizan en el nucleoplasma los ARNr se sintetizan en el nucleolo
PROCESOS PORT-TRANSCRIPCIONALES
La mayoría de los arna recién transcriptos no son definidos ni funcionales (se los llama
precursores) por lo tanto deben sufrir algún tipo de proceso a la transcripción.
ESTOS PROCESOS SON DIFERENTES EN PROCARIOTAS Y EUCARIOTAS, Y TAMBIÉN ENTRE LOS
DIVERSOS TIPOS DE ARN.
En eucariotas los procesos post-transcripcionales son:

13
I.S.U Instituto Superior Universitario
1. MADURACION DEL PRECURSOR DE LOS ARNm: CAPPING O ENCAPUCHAMIENTO,
POLIADENILACION.
2. MADURACION DEL PRECURSOR DE LOS ARNt.
3. MADURACION DELO PRECURSOR DE LOS ARNr.
Todos estos tienen lugar en el citoplasma, salvo la maduración del precursor de los ARNr, que
sucede en el núcleo.
MADURACION DEL PRECURSOR DE LOS ARNm.
Los precursores de los ARNm, son denominados transcriptos primarios, porque son copias
fieles de toda la información que contienen el gen en el ADN. Sion embargo, no toda esta
información llega al citoplasma para ser traducida, ya que ciertas porciones del transcripto son
cortadas y eliminadas (proceso conocido como Spicing) y no forman parte del ARNm maduro.
Los transcriptos primarios, además, sufren otras modificaciones en el núcleo (capping y
poliadenilación) antes de viajar al citoplasma para ser traducidos.
Splicing: el ADN de las eucariotas (y también de algunas procariotas) cuentan con
segmentos que pueden quedar representados en los ARNm maduros y otros que no.
Los que no quedan en el ARNm, son denominados INTRONES y los que pueden quedar
en el ARN son EXONES. Durante el proceso los intrones son cortados en sus extremos,
separados y luego degradados por las RNasas. A la vez, los exones se unen a otros,
formando el ARNm definitivo.
Capping: consiste en el agregado de un nucleótido inusual, en de 7-metil-guanosina-
trifosfato, en el extremo 5´de los transcriptos primarios. Este nucleótido constituye el
“CAP” o casquete o capuchón 5´que es reconocido por la subunidad menor de los
ribosomas en el proceso de traducción, y además protegería a la molécula de la acción
degradativa de las RNasas.

14
I.S.U Instituto Superior Universitario
Poliadenilación: consiste en el agregado de nucleótidos de Adenina en el extremo 3´de
los transcriptos primarios de tal manera que se forma una cola llamada POLI-A que
evitaría que la acción de las RNasas llegue a afectar a la región del transcripto primario
que contienen la información. La enzima que es la encargada de fabricar la cola POLI-A
es una polimerasa que a diferencia de las que ya vimos no necesita del ADN como
molde, sin embargo para actuar utiliza ATP.
UNA VEZ TERMINADO LOS TRES PROCESOS: CAPPING, POLIADENILACIÓN Y SPLICING, SE
OBTIENE EL ARNM LISTO PARA IR AL CITOPLASMA PARA SER TRADUCIDO.
MIENTRAS TANTO ESTÁN SIENDO MODIFICADOS LOS PRECURSORES DE LOS OTROS ARN.

15
I.S.U Instituto Superior Universitario
MADURACION DEL PRECURSOR DE LOS ARNt.
Los genes de los diferentes ARNt se encuentran repetidos y unos tras otros separados por
segmentos sin información. A esta disposición se la llama disposición Tándem. Una vez
transcriptos los percusores de los diferentes ARNt, sufren un proceso de modificación de
bases, que los provee de bases raras.
Este documento contiene más páginas...
Descargar Completo
proteinas mio.pdf
 Estamos procesando este archivo...
Estamos procesando este archivo...
 Lamentablemente la previsualización de este archivo no está disponible. De todas maneras puedes descargarlo y ver si te es útil.
Lamentablemente la previsualización de este archivo no está disponible. De todas maneras puedes descargarlo y ver si te es útil.
Descargar
 Estamos procesando este archivo...
Estamos procesando este archivo...
 Lamentablemente la previsualización de este archivo no está disponible. De todas maneras puedes descargarlo y ver si te es útil.
Lamentablemente la previsualización de este archivo no está disponible. De todas maneras puedes descargarlo y ver si te es útil.